Cromossomas em bactérias: são todos isolados e circulares?
por muitos anos, acreditava-se que todas as bactérias tinham um único cromossoma circular em comparação com os eucariotas, que têm cromossomas lineares. Talvez, isso foi devido à amostra limitada de estirpes bacterianas estudadas na época e as técnicas de sequenciamento disponíveis para examinar os cromossomas bacterianos. No entanto, esta crença foi refutada quando bactérias com cromossomas múltiplos e/ou lineares foram descobertas em 1989.
Único e Circular Cromossomo
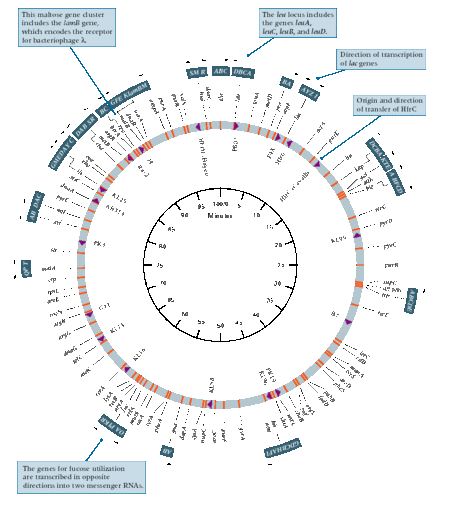
Em 1963, os estudos realizados por diversos pesquisadores determinaram que a Escherichia coli foi o genoma organizados em um único cromossoma circular . A evidência para a estrutura do DNA cromossômico foi demonstrada por imagens obtidas através de autoradiografia, microscopia eletrônica e imagens móveis de DNA usando microscopia de fluorescência. Cairns foi o primeiro pesquisador a obter uma imagem de todo o cromossomo para E. coli . A técnica utilizada foi a autoradiografia onde o cromossoma para E. coli foi rotulado usando trítio rotulado timidina, um isótopo radioativo de hidrogênio . No entanto, os tamanhos dos cromossomas eram variáveis e havia uma baixa frequência de formas circulares detectadas. Novas experiências baseadas na conjugação de Hfr, demonstrando convincentemente que a E. coli tem um cromossoma circular .
os dados publicados do cromossoma circular na E. coli foram rapidamente adoptados por muitos investigadores. Portanto, E. coli tornou-se amplamente utilizado como o modelo primário para investigar a replicação cromossômica .
Vários Cromossomos
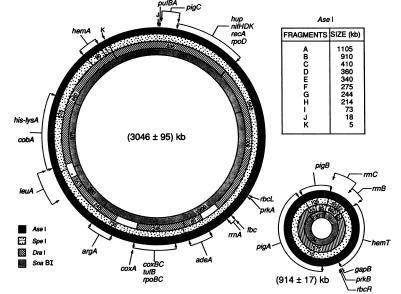
a primeira evidência de múltiplos cromossomas em bactérias foi encontrada em sphaeroides de Rodobacter, uma bactéria Gram-negativa em forma de haste (Figura 2) . Os pesquisadores foram capazes de fornecer um completo mapa físico da R. sphaeroides genoma através da obtenção de cromossômicas fragmentos de DNA através de restrição de digerir com AseI, SpeI, DraI, e SnaBI a partir do DNA genômico, que ajudou a provar a existência de vários cromossomos . O campo de pulso recebe eletroforese (PFGE) é usado para separar as moléculas de DNA através da aplicação de um campo elétrico. Ao contrário da eletroforese em gel padrão, a tensão é constantemente trocada em três direções diferentes . A suposição aqui é que o DNA circular é imóvel enquanto a maioria do DNA é mobilizado por quebra . A vantagem do PFGE é que ele pode separar DNAs de alguns quilobases (kb) para mais de 10 pares de megabase (Mb). Os fragmentos de ADN obtidos a partir da utilização de endonucleases, como o SnaBI, produzem um padrão distinto de bandas úteis para o mapeamento físico dos cromossomas. A primeira evidência que levou à ideia de dois cromossomos circulares foi da digestão do DNA genômico usando SnaBI e AseI, onde os fragmentos foram separados usando PFGE e hibridização com hemA como uma sonda . Os resultados mostraram que o fragmento de SnaBI de 784 kb estava localizado na totalidade dentro dos fragmentos asei D, E E H de 914 kb . No entanto, há um 130 kb de DNA não contabilizado, o que torna aritmeticamente impossível, uma vez que deve haver um comprimento total de fragmento de SnaBI de 1.225 kb . Portanto, os fragmentos de AseI D, E E H não podem ser unidos com os outros fragmentos de AseI contíguos . Através do processo de estreitar como os fragmentos se conectam, os cromossomos foram mapeados fisicamente. Em uma base molecular, este experimento foi usado para determinar que r. sphaeroides tem dois cromossomos circulares, onde um é de 3,1 Mb e o outro é de 0,9 Mb . Em termos de base genética, os experimentos de acasalamento interrompidos usando estirpes Hfr fortaleceram a evidência para a presença de múltiplos cromossomos em R. sphaeroides .
Linear Cromossomos
Em 1970, a primeira evidência de cromossomos lineares foi descoberto, mas a limitada disponibilidade de técnicas científicas, e uma forte crença de bactérias cromossômicas circularidade foi tão convincente que muitas pessoas não entrem nessa idéia até muito mais tarde. Em 1989, a técnica de eletroforese em gel de campo pulsado foi desenvolvida, e usada em conjunto com digestões de restrição para verificar que a espiroqueta Borrelia burgdorferi tem um cromossomo linear usando um processo similar de estreitamento, conectando os fragmentos separados como os r. sphaeroides . B. burgdorferi foi a primeira bactéria a mostrar que os cromossomos lineares estão presentes nos procariontes, e o tamanho do cromossomo foi encontrado em torno de 1,0 Mb . Esta organização cromossómica é comparável à organização cromossómica eucariótica.
problemas com cromossomas Lineares
dois problemas surgem com a presença e o uso de cromossomas lineares em comparação com cromossomas circulares em procariontes. Em primeiro lugar, as extremidades livres de ADN de cadeia dupla devem ter algum tipo de protecção para que as nucleases intracelulares não as degradem . Em segundo lugar, os telómeros, que são os fins das moléculas lineares de ADN, necessitarão de um processo diferente para a replicação do ADN . Para proteção, existem dois tipos de telômeros que foram observados. O primeiro é chamado de palindromic hairpin loops, em que não há nenhuma extremidade dupla livre disponível,. O outro tipo é a telómeros invertron onde os cromossomas contêm uma proteína que se liga às extremidades de 5′. Isto pode ser encontrado em cromossomas lineares Streptomyces. Em termos de replicação de DNA das extremidades das moléculas lineares de DNA, não se sabe muito sobre o mecanismo.
outros resultados de cromossomas Lineares
s. griseus é uma bactéria Gram-positiva conhecida pela produção do antibiótico, a estreptomicina . Esta bactéria também tem um cromossoma linear identificado através de eletroforese em gel de campo pulsado em conjunto com digestões de restrição . Existem também outras espécies de Streptomyces com cromossomas lineares identificados.
Agrobacterium tumefaciens
Usando eletroforese em gel de campo pulsado, o genoma de A. tumefaciens foi analisado, onde um cromossoma linear e um cromossoma circular foram identificados . O tamanho do cromossomo linear é de 2100 Kb, que é menor que o cromossomo circular de 3000 Kb . A espécie de Agrobacterium tem uma relação próxima de sequência rRNA 16s com a espécie Brucella, que tem membros conhecidos por ter dois cromossomos circulares .
outros resultados de cromossomas múltiplos
b. melitensis é uma bactéria cocobacillus Gram-negativa, com dois cromossomas circulares . Os dois tamanhos do cromossomo são 2117 Kb e 1178 Kb . Outras espécies Brucelas com dois cromossomas circulares incluem B. suis biovar 1, B. suis biovar 2, B. suis biovar 4, B. abortus e B. ovis .
Paracoccus denitrificans
P. denitrificans é uma bactéria especial com três cromossomas circulares identificados pela electroforese em gel de impulso . Os tamanhos aproximados dos cromossomos são 2, 1,1 e 0,64 Mb .
Vibrio Species
Existem muitas outras bactérias com múltiplos cromossomas, mas as espécies de Vibrio têm membros que têm dois cromossomas circulares . Estes incluem V. cholerae, v. parahaemolyticus, V. vulnificus e V. fluvialis .
Summary of Currently Known Bacterial Chromosome Organizations
| Bacteria | Chromosome Organization |
|---|---|
| Agrobacterium tumefaciens | One linear and one circular |
| Bacillus subtilis | Single and circular |
| Bacillus subtilis | Single and linear |
| Borrelia burgdorferi | Two circular |
| Brucella abortus | Two circular |
| Brucella melitensis | Two circular |
| Brucella ovis | Two circular |
| Brucella suis biovar 1 | Two circular |
| Brucella suis biovar 2 | Two circular |
| Brucella suis biovar 4 | Two circular |
| Escherichia coli | Single and circular |
| Paracoccus denitrificans | Three circular |
| Pseudomonas aeruginosa | Single and circular |
| Rhodobacter sphaeroides | Two circular |
| Streptomyces griseus | Linear |
| Vibrio cholerae | Two circular |
| Vibrio fluvialis | Two circular |
| Vibrio parahaemolyticus | Two circular |
| Vibrio vulnificus | Two circular |
(1) Allardet-Servent, A., Michaux-Charachon, S., Jumas-Bilak, E., Karayan, L. and Ramuz, M. “Presence of One Linear and One Circular Chromosome in the Agrobacterium tumefaciens C58 Genome.” Journal of Bacteriology, 1993, 175(24): 7869-7874
(2) Baril, C., Richaud, C., Baranton, G. and Saint-Girons, I. “Linear Chromosome of Borrelia burgdorferi.”Res. Microbiol., 1989, 140: 507-516
(3) Bendich, A. J. “The Form of Chromosomal DNA Molecules in Bacterial Cells.”Biochimie, 2001, 83: 177-186
(4) Cairns, J. “The Bacterian Chromosome and its Manner of Replication As Seen By Autoradiography.”J. Mol. Biol., 1963, 6: 208-213
(5) Del Vecchio, V. G., Kapatral, V., Elzer, P., Patra, G. and Mujer, C. V. “The Genome of Brucella melitensis.”Veterinária microbiologia, 2002, 90: 587-592
(6) Egan, E. S., Fogel, M. A. e Waldor, M. K. “Dividido Genomas: A negociar o ciclo celular em procariontes com vários cromossomas.”Microbiologia Molecular, 2005, 56(5): 1129-1138
(7) Griswold, A. “Genome Packaging in Procaryotes: the Circular Chromosome of E. coli.”Nature Education, 2008, 1 (1)
(8) Hinnebusch, J. and Tilly, K. “Linear Plasmids and Chromosomes in Bacteria.”Microbiologia Molecular, 1993, 10(5): 917-922
(9) Jumas-Bilak, E., Michaux-Charachon, S., Bourg, G., O’Callaghan, D. and Ramuz, M. “Differences in Chromosome Number and Genome Rearrangements in the genome Brucella.” Mol. Microbiol., 1998, 27(1): 99-106
(10) Lezhava A., Mizukami, T., Kajitani, T., Kameoka, D., Redenbach, M., Shinkawa, H., Nimi, O. e Kinashi, H. “Mapa Físico do Cromossomo Linear de Stremptomyces griseus.”Journal of Bacteriology, 1995, 177(22): 6492-6498
(11) Suwanto, A. and Kaplan, S. “Chromosome Transfer in Rhodobacter sphaeroides: HFR Formation and Genetic Evidence for Two Unique Circular Chromosomes.”Journal of Bacteriology, 1992, 174(4): 1135-1145
(12) Suwanto, A. and Kaplan, S. “Physical and Genetic Mapping of the Rhodobacter sphaeroides 2.4.1 Genome: Presença de dois cromossomas circulares únicos.”Journal of Bacteriology, 1989, 171(11): 5850-5859
(13) Ussery, D. “Bacteriophage Genetics.”2000, http://www.cbs.dtu.dk/staff/dave/roanoke / genetics980311.html
(14) Volff, J. N. and Altenbuchner, J. “A New Beginning with New Ends: Linearisation of Circular Chromosomes during Bacterial Evolution.”MEMS Microbiology Letters, 2000, 186: 143-150
(15) Winterstein, C. and Ludwig, B. “Genes Coding for Respiratory Complexes Map on All Three Chromosomes of the Paracoccus denitrificans Genome.” Arco. Microbiol., 1998, 169(4): 275-281
(16) Yamaichi, Y., Iida, T., Park, K. S., Yamamoto, K. and Honda, T. “Physical and Genetic Map of the Genome of Vibrio parahaemolyticus: Presence of Two Chromosomes in Vibrio Species.”Mol Microbiol., 1999, 31(5): 1513-1521
Leave a Reply